class: center, middle, inverse, title-slide # Numpy ### Licenciatura en Ciencias Genómicas, UNAM ### First version: 2021-08-22; Last update: 2021-10-19 --- <style type="text/css"> /* From https://github.com/yihui/xaringan/issues/147 */ .scroll-output { height: 80%; overflow-y: scroll; } /* https://stackoverflow.com/questions/50919104/horizontally-scrollable-output-on-xaringan-slides */ pre { max-width: 100%; overflow-x: scroll; } </style> <iframe width="560" height="315" src="https://www.youtube.com/embed/RdUjDbuIxjg" title="YouTube video player" frameborder="0" allow="accelerometer; autoplay; clipboard-write; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> --- <img src="imgs/clase_5/array_dim.png" width="800px" style="display: block; margin: auto;" /> --- ```python import numpy as np array_1D = np.array([1,2,3]) array_1D ``` ``` ## array([1, 2, 3]) ``` ```python array_2D = np.array([ [1,2,3], (2,3,4)]) array_2D ``` ``` ## array([[1, 2, 3], ## [2, 3, 4]]) ``` ```python array_3D = np.array([ [ [1, 2], [3, 4] ], [ [5, 6], [7, 8] ] ]) array_3D ``` ``` ## array([[[1, 2], ## [3, 4]], ## ## [[5, 6], ## [7, 8]]]) ``` --- <iframe width="560" height="315" src="https://www.youtube.com/embed/FpkG7KPfd9c" title="YouTube video player" frameborder="0" allow="accelerometer; autoplay; clipboard-write; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> --- <img src="imgs/clase_5/array_1D.png" width="800px" style="display: block; margin: auto;" /> --- Creamos un array ```python import numpy as np # biomasa en unidades de absorbancia ecoli_matraz = np.array([0.1, 0.15, 0.19, 0.5, 0.9, 1.4, 1.8, 2.1, 2.3]) ecoli_matraz.ndim ``` ``` ## 1 ``` ```python ecoli_matraz.shape ``` ``` ## (9,) ``` ```python len(ecoli_matraz) ``` ``` ## 9 ``` --- <img src="imgs/clase_5/array_2D.png" width="800px" style="display: block; margin: auto;" /> --- ```python # Biomasa en unidades de absorbancia (OD600) ecoli_m_b = np.array([[0.1, 0.15, 0.19, 0.5, # Matraz 250 mL 0.9, 1.4, 1.8, 2.1, 2.3], [0.1, 0.17, 0.2, 0.53, # Biorreactor 50 L 0.97, 1.43, 1.8, 2.1, 2.8], [0.1, 0.17, 0.2, 0.52, # B. alimentado 50 L 0.95, 1.41, 1.8, 2.2, 2.8] * ]) ecoli_m_b.ndim ``` ``` ## 2 ``` ```python ecoli_m_b.shape ``` ``` ## (3, 9) ``` ```python len(ecoli_m_b) ``` ``` ## 3 ``` --- Una `\(OD_{600}\)` de 1 representa 0.39 `\(g/L\)` de peso seco. ¿Cómo lo harían? -- ```python ecoli_matraz # OD600 ``` ``` ## array([0.1 , 0.15, 0.19, 0.5 , 0.9 , 1.4 , 1.8 , 2.1 , 2.3 ]) ``` -- ```python ecoli_matraz_gL = ecoli_matraz*0.39 ecoli_matraz_gL ``` ``` ## array([0.039 , 0.0585, 0.0741, 0.195 , 0.351 , 0.546 , 0.702 , 0.819 , ## 0.897 ]) ``` --- # Operaciones ### Suma, resta, multiplicación, división Tengo 2 bacterias produciendo 2 metabolitos de interés biotecnológico. Si al final de mi producción en un biorreactor con 50 L tengo las siguientes cantidades de metabolito en g/L: | | **Metabolito A** | **Metabolito B** | |:--------:|:---------:|:---------:| | Bacteria 1 | 16 | 14 | | Bacteria 2 | 12 | 9 | ¿Cuál sería el total de mi producción si tengo 2 biorreactores? --- ```python produccion = np.array([[16, 14], [12, 9]]) produccion ``` ``` ## array([[16, 14], ## [12, 9]]) ``` -- ```python produccion+produccion # Por bacteria y metabolito ``` ``` ## array([[32, 28], ## [24, 18]]) ``` -- ```python np.sum(produccion*2, axis= 0) # Por metabolito ``` ``` ## array([56, 46]) ``` -- ```python np.sum(produccion*2, axis= 1) # Por bacteria ``` ``` ## array([60, 42]) ``` --- Al extraer el producto, se me contaminó la mitad del líquido de uno de los biorreactores ```python total = produccion*2 total ``` ``` ## array([[32, 28], ## [24, 18]]) ``` -- ```python contaminado = produccion/2 contaminado ``` ``` ## array([[8. , 7. ], ## [6. , 4.5]]) ``` -- ```python total_real = total - contaminado total_real ``` ``` ## array([[24. , 21. ], ## [18. , 13.5]]) ``` --- Para crear un fármaco, necesito las siguientes cantidades de los metabolitos de cada bacteria (ya que los metabolitos presentan diferentes glicosilaciones): | | **Metabolito A** | **Metabolito B** | |:--------:|:---------:|:---------:| | Consumo bac1 | 7 | 3 | | Consumo bac2 | 5 | 2 | ¿Cuánto producto me sobra ? ```python consumo = np.array([[7, 3],[5, 2]]) total_real ``` ``` ## array([[24. , 21. ], ## [18. , 13.5]]) ``` ```python total_real-consumo ``` ``` ## array([[17. , 18. ], ## [13. , 11.5]]) ``` --- ¿Si se necesitara la misma cantidad por cada bacteria? ¿Cómo crearian ahora a la variable consumo? -- ```python consumo = np.array([[7, 3],[7, 3]]) total_real - consumo # Por bacteria ``` ``` ## array([[17. , 18. ], ## [11. , 10.5]]) ``` -- Al igual que en R, podemos reciclar: ```python consumo = np.array([7, 3]) total_real - consumo # Por bacteria ``` ``` ## array([[17. , 18. ], ## [11. , 10.5]]) ``` --- # Otras funciones Potencias ```python total_real ``` ``` ## array([[24. , 21. ], ## [18. , 13.5]]) ``` ```python total_real**2 ``` ``` ## array([[576. , 441. ], ## [324. , 182.25]]) ``` ```python total_real**total_real ``` ``` ## array([[1.33373578e+33, 5.84258702e+27], ## [3.93464081e+22, 1.81763164e+15]]) ``` --- # Otras funciones Transpuesta y suma total ```python total_real ``` ``` ## array([[24. , 21. ], ## [18. , 13.5]]) ``` ```python total_real.T ``` ``` ## array([[24. , 18. ], ## [21. , 13.5]]) ``` ```python total_real.sum() ``` ``` ## 76.5 ``` --- # Otras funciones Mínimo y máximo ```python total_real ``` ``` ## array([[24. , 21. ], ## [18. , 13.5]]) ``` ```python total_real.min() ``` ``` ## 13.5 ``` ```python print( total_real.max(), np.max(total_real) ) ``` ``` ## 24.0 24.0 ``` --- # Otras funciones Exponencial y raíz cuadrada ```python total_real ``` ``` ## array([[24. , 21. ], ## [18. , 13.5]]) ``` ```python np.exp(total_real) ``` ``` ## array([[2.64891221e+10, 1.31881573e+09], ## [6.56599691e+07, 7.29416370e+05]]) ``` ```python np.sqrt(total_real) ``` ``` ## array([[4.89897949, 4.58257569], ## [4.24264069, 3.67423461]]) ``` --- # Otras funciones Trigonométricas ```python np.sin(np.array([np.pi, np.pi/2])) ``` ``` ## array([1.2246468e-16, 1.0000000e+00]) ``` ```python np.arcsin(np.array([0.0, 1.0])) ``` ``` ## array([0. , 1.57079633]) ``` --- # Otras funciones Calcular y asignar ```python total_real ``` ``` ## array([[24. , 21. ], ## [18. , 13.5]]) ``` ```python total_real += 2 total_real ``` ``` ## array([[26. , 23. ], ## [20. , 15.5]]) ``` ```python total_real *= 2 total_real ``` ``` ## array([[52., 46.], ## [40., 31.]]) ``` --- # Otras funciones Redondeo ```python redondear = np.array([1.1, 1.5, 1.9, 2.5]) np.floor(redondear) ``` ``` ## array([1., 1., 1., 2.]) ``` ```python np.ceil(redondear) ``` ``` ## array([2., 2., 2., 3.]) ``` ```python np.round(redondear) ``` ``` ## array([1., 2., 2., 2.]) ``` --- # <span style="color:Plum">Ejercicio 1</span> Al inducir 4 genes de producción a diferentes temperaturas se obtuvieron las siguientes producciones del metabolito de interés en g/L: | | **30 °C** | **35 °C** | |:--------:|:---------:|:---------:| | Gen 1 | 5 | 3 | | Gen 2 | 11 | 7 | | Gen 3 | 4 | 9 | | Gen 4 | 2 | 6 | Cada gen tiene un inductor diferente y cada uno tuvo los siguientes costos: | | **Costo de inducción** | |:-----:|:----------------------:| | Gen 1 | 3.5 | | Gen 2 | 5 | | Gen 3 | 7 | | Gen 4 | 4.3 | **¿Qué gen nos conviene inducir y a qué temperatura para obtener nuestro metabolito?** --- ```python produccion = np.array([ [5,3], [11, 7], [4, 9], [2, 6]]) produccion ``` ``` ## array([[ 5, 3], ## [11, 7], ## [ 4, 9], ## [ 2, 6]]) ``` ```python costos = np.array([3.5, 5, 7, 4.3]) costos ``` ``` ## array([3.5, 5. , 7. , 4.3]) ``` --- ### Tipos de datos `dtype` ```python from sys import getsizeof *np_float = np.array([1.0, 2.0, 3.0, 4.0]) print("Tipo de dato\t", np_float.dtype, "\nTamaño en bytes\t", getsizeof(np_float)) ``` ``` ## Tipo de dato float64 ## Tamaño en bytes 128 ``` ```python *np_int = np.array([1, 2, 3, 4]) print("Tipo de dato\t", np_int.dtype, "\nTamaño en bytes\t", getsizeof(np_int)) ``` ``` ## Tipo de dato int32 ## Tamaño en bytes 112 ``` --- También podemos especificar el tipo de dato ```python np_float = np.array([1, 2, 3, 4], * dtype='float64') print("Tipo de dato\t", np_float.dtype, "\nTamaño en bytes\t", getsizeof(np_float)) ``` ``` ## Tipo de dato float64 ## Tamaño en bytes 128 ``` ```python np_int = np.array([1.0, 2.0, 3.0, 4.0], * dtype='int32') print("Tipo de dato\t", np_int.dtype, "\nTamaño en bytes\t", getsizeof(np_int)) ``` ``` ## Tipo de dato int32 ## Tamaño en bytes 112 ``` --- Tipos de datos: https://numpy.org/doc/stable/reference/arrays.dtypes.html#arrays-dtypes + Tipo de dato booleano ```python bool_np = np.array([True, False, True, False]) bool_np.dtype ``` ``` ## dtype('bool') ``` -- Accedemos con el array booleano ```python np_int ``` ``` ## array([1, 2, 3, 4]) ``` ```python np_int[bool_np] ``` ``` ## array([1, 3]) ``` --- Otros ejemplos de acceso con booleanos ```python np_int ``` ``` ## array([1, 2, 3, 4]) ``` ```python np_int <3 ``` ``` ## array([ True, True, False, False]) ``` ```python np_int[np_int <3] ``` ``` ## array([1, 2]) ``` --- Otros ejemplos de acceso con booleanos ```python np_int ``` ``` ## array([1, 2, 3, 4]) ``` ```python np_int.max() ``` ``` ## 4 ``` ```python np_int[np_int == np_int.max()] ``` ``` ## array([4]) ``` --- Otros ejemplos de acceso con booleanos ```python np_int ``` ``` ## array([1, 2, 3, 4]) ``` ```python (np_int <2 ) | (np_int >3) ``` ``` ## array([ True, False, False, True]) ``` ```python np_int[(np_int <2 ) | (np_int >3)] ``` ``` ## array([1, 4]) ``` --- # <span style="color:Plum">Ejercicio 2</span> Del ejericio anterior, imprime el costo de producción más alto y el más bajo utilizando booleanos. --- + Tipo de dato complejo ```python num_1 = np.array([3+6j]) num_2 = np.array([7+2j]) num_1.dtype ``` ``` ## dtype('complex128') ``` ```python num_1.real ``` ``` ## array([3.]) ``` ```python num_1.imag ``` ``` ## array([6.]) ``` ```python num_1+num_2 ``` ``` ## array([10.+8.j]) ``` --- + Tipo de dato fecha: `ISO 8601` o formato `datetime` ```python dias = np.datetime64('2005-02-25') dias.dtype ``` ``` ## dtype('<M8[D]') ``` ```python meses = np.datetime64('2005-02') meses.dtype ``` ``` ## dtype('<M8[M]') ``` ```python forzar_dias = np.datetime64('2005-02', 'D') forzar_dias.dtype ``` ``` ## dtype('<M8[D]') ``` --- Comparar fechas ```python np.datetime64('2005') == np.datetime64('2005-01-01') ``` ``` ## True ``` Cálculos con fechas ```python np.datetime64('2009-01-01') - np.datetime64('2008-01-01') ``` ``` ## numpy.timedelta64(366,'D') ``` ```python np.datetime64('2009') + np.timedelta64(20, 'D') ``` ``` ## numpy.datetime64('2009-01-21') ``` --- # Acceder al array 1D ```python ecoli_matraz ``` ``` ## array([0.1 , 0.15, 0.19, 0.5 , 0.9 , 1.4 , 1.8 , 2.1 , 2.3 ]) ``` ```python ecoli_matraz[2] ``` ``` ## 0.19 ``` ```python ecoli_matraz[2:5] ``` ``` ## array([0.19, 0.5 , 0.9 ]) ``` ```python ecoli_matraz[0:6:2] # Del 0 al 6 de 2 en 2 ``` ``` ## array([0.1 , 0.19, 0.9 ]) ``` --- ```python ecoli_matraz ``` ``` ## array([0.1 , 0.15, 0.19, 0.5 , 0.9 , 1.4 , 1.8 , 2.1 , 2.3 ]) ``` ```python ecoli_matraz[1:7:2] ``` ``` ## array([0.15, 0.5 , 1.4 ]) ``` ```python ecoli_matraz[:6:2] # Del 0 al 6 de 2 en 2 ``` ``` ## array([0.1 , 0.19, 0.9 ]) ``` ¿Qué creen que harán las siguientes líneas? + ecoli_matraz[::2] + ecoli_matraz[1::2] + ecoli_matraz[::-1] --- ```python ecoli_matraz[::2] ``` ``` ## array([0.1 , 0.19, 0.9 , 1.8 , 2.3 ]) ``` ```python ecoli_matraz[1::2] ``` ``` ## array([0.15, 0.5 , 1.4 , 2.1 ]) ``` ```python ecoli_matraz[::-1] ``` ``` ## array([2.3 , 2.1 , 1.8 , 1.4 , 0.9 , 0.5 , 0.19, 0.15, 0.1 ]) ``` ```python ecoli_matraz[-1] ``` ``` ## 2.3 ``` --- ## Acceder al array 2D ```python produccion ``` ``` ## array([[ 5, 3], ## [11, 7], ## [ 4, 9], ## [ 2, 6]]) ``` ```python print(produccion[2],'\n', produccion[2:4]) ``` ``` ## [4 9] ## [[4 9] ## [2 6]] ``` ```python produccion[0:6:2] # Del 0 al 6 de 2 en 2 ``` ``` ## array([[5, 3], ## [4, 9]]) ``` --- ```python produccion ``` ``` ## array([[ 5, 3], ## [11, 7], ## [ 4, 9], ## [ 2, 6]]) ``` ```python produccion[2][1] ``` ``` ## 9 ``` ```python produccion[2,1] ``` ``` ## 9 ``` ¿Será igual `produccion[0:6:2][1]` que `produccion[0:6:2, 1]` ? -- ```python produccion[0:6:2][1] , produccion[0:6:2, 1] ``` ``` ## (array([4, 9]), array([3, 9])) ``` --- ```python produccion ``` ``` ## array([[ 5, 3], ## [11, 7], ## [ 4, 9], ## [ 2, 6]]) ``` ```python produccion[-2:,-2:] ``` ``` ## array([[4, 9], ## [2, 6]]) ``` ```python produccion[::2,1::2] ``` ``` ## array([[3], ## [9]]) ``` --- # ... x[1, 2, ...] es x[1, 2, :, :, :], x[..., 7] es x[:, :, :, :, 7] x[4, ..., 5, :] es x[4, :, :, 5, :] ```python a_3D = np.array([[[ 1, 2, 3], [ 11, 12, 13]], [[101, 102, 103], [1001, 1002, 1003]]]) a_3D.shape ``` ``` ## (2, 2, 3) ``` ```python a_3D[1, ...] # a_3D[1, :, :] o a_3D[1] ``` ``` ## array([[ 101, 102, 103], ## [1001, 1002, 1003]]) ``` --- ```python a_3D ``` ``` ## array([[[ 1, 2, 3], ## [ 11, 12, 13]], ## ## [[ 101, 102, 103], ## [1001, 1002, 1003]]]) ``` ```python a_3D[..., 2] # a_3D[:, :, 2] ``` ``` ## array([[ 3, 13], ## [ 103, 1003]]) ``` --- **Funciones para crear arrays** arange y linspace ```python np.arange(10) ``` ``` ## array([0, 1, 2, 3, 4, 5, 6, 7, 8, 9]) ``` ```python np.arange(0, 10, 2) ``` ``` ## array([0, 2, 4, 6, 8]) ``` ```python np.linspace(0, 8, 5) ``` ``` ## array([0., 2., 4., 6., 8.]) ``` ¿Qué creen que nos dé `np.arange(0.5, 0.8, 0.1)` -- ```python np.arange(0.5, 0.8, 0.1) ``` ``` ## array([0.5, 0.6, 0.7, 0.8]) ``` --- ```python np.arange(0.5, 0.75, 0.1) ``` ``` ## array([0.5, 0.6, 0.7]) ``` ```python np.linspace(0.5,0.7, 3) ``` ``` ## array([0.5, 0.6, 0.7]) ``` --- Random ```python np.random.randint(0, 10, 3) ``` ``` ## array([5, 0, 1]) ``` ```python np.random.rand(3) ``` ``` ## array([0.28326277, 0.88271753, 0.5991886 ]) ``` ```python np.random.uniform(1, 10, 3) ``` ``` ## array([8.87159061, 8.38506923, 8.00215397]) ``` ```python np.random.normal(5, 2, 3) ``` ``` ## array([5.86358535, 4.61688963, 6.47040085]) ``` ```python np.random.poisson(10, 3) ``` ``` ## array([12, 5, 11]) ``` --- Ceros, unos, diagonales ```python np.zeros((2,3)) ``` ``` ## array([[0., 0., 0.], ## [0., 0., 0.]]) ``` ```python np.ones((3,3)) ``` ``` ## array([[1., 1., 1.], ## [1., 1., 1.], ## [1., 1., 1.]]) ``` ```python np.eye(3) ``` ``` ## array([[1., 0., 0.], ## [0., 1., 0.], ## [0., 0., 1.]]) ``` --- También se puede: + Repetir + Unir (como cbind y rbind en R ) + Dividir + Borrar filas o columnas + Insertar filas o columnas + Ordenar por filas o columnas + Cambiar la forma --- # Diferencias array y lista python https://www.bioinformaticscrashcourse.com/7_DataAnalysisWithPython.html 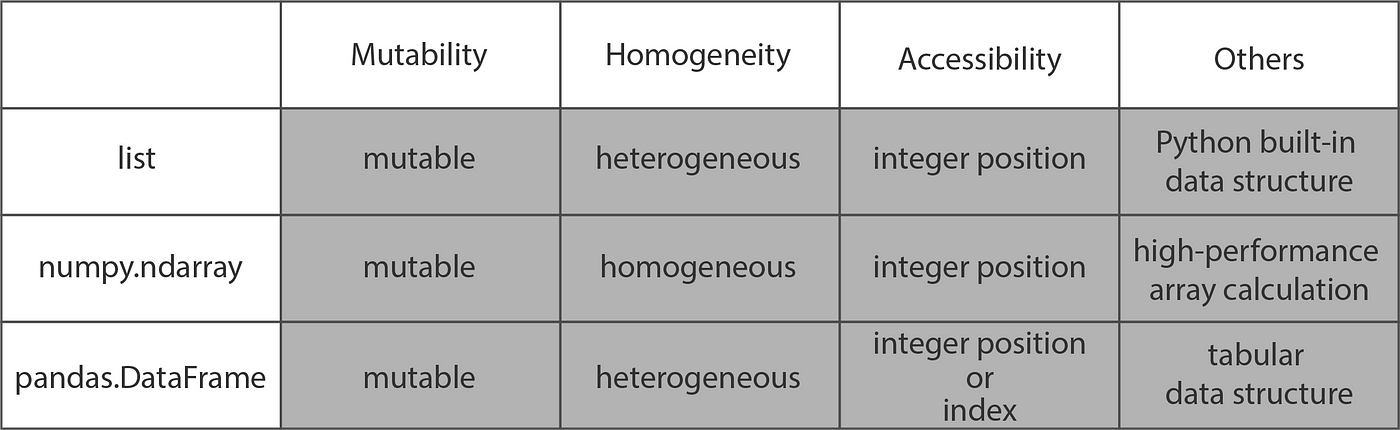 ```python ej_lista = [[1,3,4],[6,9,11]] ej_array = np.array(ej_lista) #ej_lista[1,2] ej_array[1,2] ``` ``` ## 11 ``` --- ```python ej_lista = [[0,0,0]]*2 ej_lista[0][0] =3 ``` ¿Qué nos va a arrojar? -- ```python ej_lista ``` ``` ## [[3, 0, 0], [3, 0, 0]] ``` ```python ej_array = np.array([[0,0,0]]) ej_array = np.tile(ej_array,(2,1)) ej_array[0][0] = 3 ej_array ``` ``` ## array([[3, 0, 0], ## [0, 0, 0]]) ``` --- ```python ej_array = np.array([[0]*3]*2) ej_array[0][0] = 73 ej_array ``` ``` ## array([[73, 0, 0], ## [ 0, 0, 0]]) ``` --- # Array estructurado ```python mascotas = np.array([('Freya', 6, 6.5), ('Senna', 1, 2.5)], dtype=[('nombre', (np.str_, 10)), ('edad', np.int32), ('peso', np.float64)]) mascotas ``` ``` ## array([('Freya', 6, 6.5), ('Senna', 1, 2.5)], ## dtype=[('nombre', '<U10'), ('edad', '<i4'), ('peso', '<f8')]) ``` ```python sort_age = np.sort(mascotas, order='edad') sort_name = np.sort(mascotas, order='nombre') sort_age ``` ``` ## array([('Senna', 1, 2.5), ('Freya', 6, 6.5)], ## dtype=[('nombre', '<U10'), ('edad', '<i4'), ('peso', '<f8')]) ``` ```python sort_name ``` ``` ## array([('Freya', 6, 6.5), ('Senna', 1, 2.5)], ## dtype=[('nombre', '<U10'), ('edad', '<i4'), ('peso', '<f8')]) ``` --- # <span style="color:OrangeRed">Tarea</span> Crear arrays estructurados de los arrays creados en el ejercicio 1.